ACHTUNG: diese Seite ist archiviert und wird NICHT MEHR von den automatischen Prozessen aktualisiert. Diese Seite hier wird nur noch aus „historischem Interesse“ angeboten.
Wie breitet sich die Omikron Variante und ihre Untertypen BA1 und BA2 in Deutschland aus? Hier wird die Entwicklung anhand von Grafiken gezeigt. Basis sind dabei die vom RKI gesammelten Sequenzierungen. Diese Seite wird täglich aktualisiert. Schauen sie also häufiger vorbei. Eine Übersicht über die einzelnen Varianten findet sich hier.

Grafik nach:
Darstellung:
Anzahl Tage:
Anlass für Sequenzierung:
Varianten:
Andere
Alpha
Delta
Omicron
[
BA.1
BA.2
BA.3
BA.4
BA.5
]
Hospitalisierungen werden häufig im Zusammenhang mit Virusvarianten genannt. Hier geht es zu einer entsprechenden Darstellung der Hospitalisierungsinzidenz.
Ich werde häufig nach Daten pro Bundesland gefragt. Aktuell rechne ich das nicht, da die Sequenzzahlen der repräsentativen Stichprobe schon bundesweit sehr gering sind. Ich habe gewisse Zweifel an der Aussagekraft, wenn man das noch auf die Länder herunter bricht. Allerdings überlege ich, künftig doch einmal einen Versuch zu machen, gerade um die Problematik genauer aufzeigen zu können. Das braucht allerdings Zeit, die ich aktuell nicht habe.

Wachstum der Corona-Varianten in Deutschland
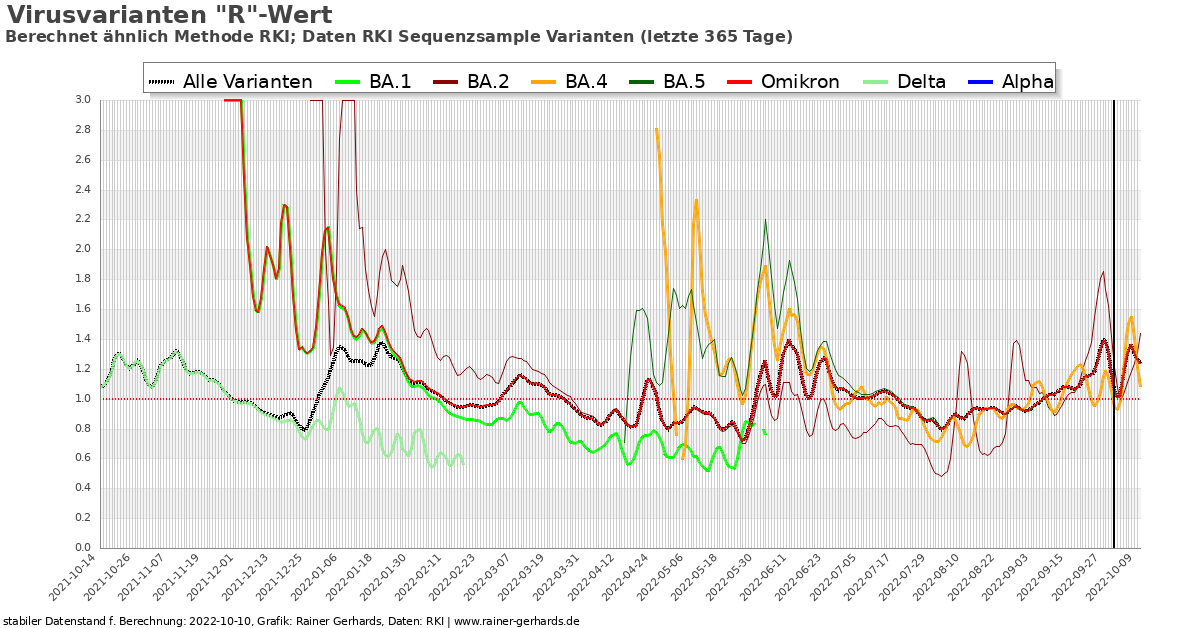
Achtung: die letzten 10 Tage sind unvollständig und können sich noch stark ändern.
Varianten mit einem Anteil von unter einem Promille zum jeweiligen Zeitpunkt werden in der Grafik nicht angezeigt. Dies würde nur zu irritierenden und nicht aussagekräftigen Ausschlägen führen. In Bezug auf neue Varianten kann sich hierdurch eine nur gering zeitlich Verzögerung bis zu deren Anzeige ergeben.
Wie wächst Omicron, BA.1, BA.2 in absoluten Zahlen?
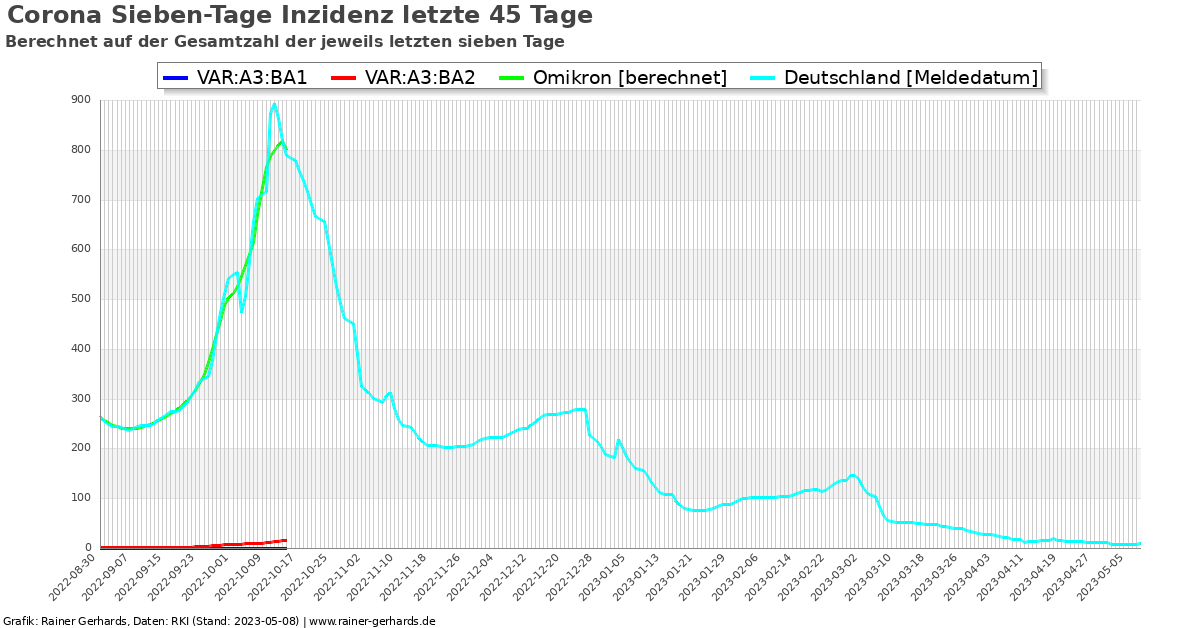
Die Hochrechnung der absoluten Varianten-Fallzahl anhand der täglichen Neuinfektionen ist lediglich nach Meldedatum sinnvoll. Die Berechnung anhand des Eingangsdatums RKI ist wenig sinnvoll, da in der Regel Sequenzen mehrerer Tage zusammentreffen und darüber hinaus die exakten Referenzdaten nicht hinreichend klar zuzuordnen sind. Auch Berechnungsversuche zeigten die Ungeeignetheit dieser Methode.

Die Daten werden tendenziell täglich aktualisiert und Grafiken fortgeschrieben. Auch das kann am Anfang aber evtl. noch nicht 100% funktionieren. Ich plane, das Ganze noch weiter zu entwickeln. Anregungen sind ebenfalls willkommen.
Eine lesenswerte Quelle ist die „Tägliche Übersicht zu Omikron-Fällen“ das RKI!
Aktualität der Sequenzdaten

Die Sequenzdatenbank hat einen gewissen zeitlichen Versatz. Grob ist der Ablauf wie folgt:
- Die Probe wird für den PCR-Test entnommen
- Die Probe wird zum Labor gesendet, ggf. wird sie von dort (bei Überlastung) an ein weiteres Labor gesendet
- PCR wird durchgeführt, es wird entschieden die Probe zu sequenzieren
- die Probe wird sequenziert
- die Sequenzen werden dem RKI gemeldet, eine erste publikation auf github erfolgt
- das RKI untersucht und klassifiziert die Sequenz
- Sequenzen, die eindeutig klassifizierbar waren, werden in den Datensatz der Entwicklungslinien (lineages) eingefügt
- der Datensatz Entwicklungslinien wird auf github publiziert
Der gesamte Prozess benötigt üblicherweise zwischen 10 und 15 Tagen. Die Grafik oben visualisiert die Menge der Sequenzen, die noch nicht abschließend klassifiziert sind sowie die tägliche Zu- und Abnahme dieser Sequenzen. Zunahmen erfolgen nach Eingang neuer Daten beim RKI, Abgänge nach Klassifizierung und Aufnahme in „Entwicklungslinien“. Die Gesamtmenge wächst an, da immer ein gewisser Teil grundsätzlich nicht klassifizierbar ist (z.B. Qualitätsmängel).
Idealerweise ergeben sich keine täglichen Schwankungen. Denn dann werden eingehende Proben noch am selben Tag klassifiziert. In der Praxis gibt es aber deutliche Verzögerungen, und auch Tage ganz ohne Meldung. Dies visualisieren die blauen Balken in der Grafik. Man darf vermuten, dass die aktuell gemeldeten Daten desto unvollständiger sind, desto unregelmäßiger die tägliche Veränderung der Werte ist.
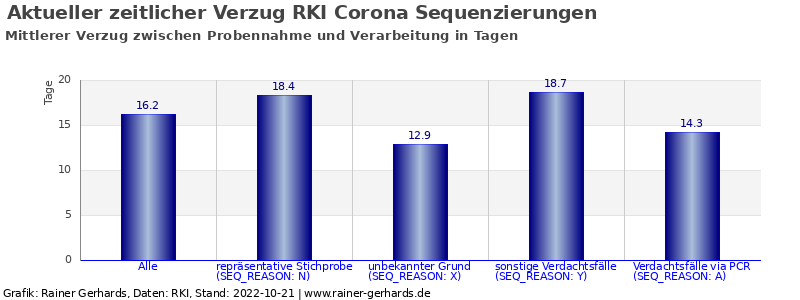
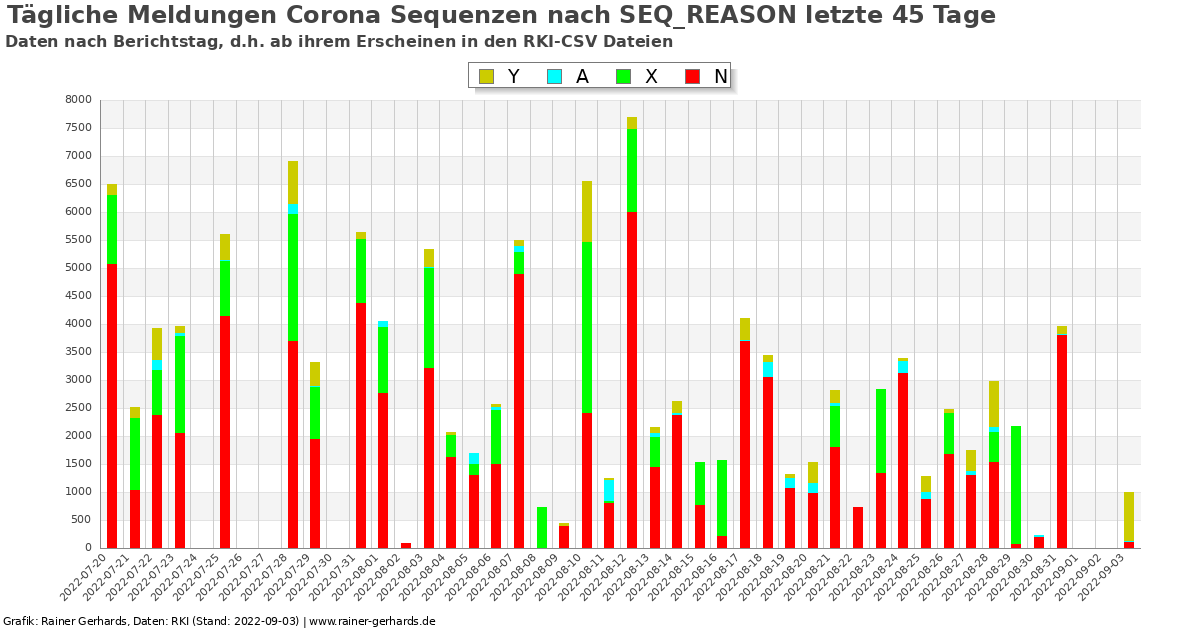
Experimentelle Auswertungen
Auf Wunsch zeigen wir hier auch einige experimentelle Auswertungen. Diese sind zwar „im großen und ganzen“ korrekt, können aber noch leichte Abweichungen zur exakten Datenlage haben. Außerdem sind theoretisch auch größere Probleme möglich, da sie nicht vollständig qualitätsgesichert sind. Nutzen Sie diese also mit Vorsicht!


Datensätze zum Download
Viele Datensätze sind zum Download und zur Weiterverarbeitung verfügbar. Bitte schauen Sie auf unserer OpenData Seite nach!
Weiters ist eine detaillierte Beschreibung der von uns verwendeten Berechnungsmethoden verfügbar.
Nützliche weitere Links
Quellen
- RKI Sequenzdaten Repository auf github
- Beschreibung der Bedeutung der Metadaten (RKI)
- Beschreibung der PANGO-Lineages mit Subvarianten (zum Abgleich von Lineage erforderlich)
Danksagungen
Einen herzlichen Dank an das RKI aber insbesondere auch Cornelius Römer, der mich auf die neue Datensammlung aufmerksam gemacht hat. Ein weiterer Dank an Ralph Einfeldt, der mich auf die konkrete Bedeutung das Datenfelds SEQ_REASON hingewiesen hat.
